paper: https://arxiv.org/abs/2102.04306
TransUNet: Transformers Make Strong Encoders for Medical Image Segmentation
Medical image segmentation is an essential prerequisite for developing healthcare systems, especially for disease diagnosis and treatment planning. On various medical image segmentation tasks, the u-shaped architecture, also known as U-Net, has become the
arxiv.org
TransUNet은 UNet의 이점과 Transformer의 이점을 가지고 있는 medical image segmentaion model이다.
- UNet - CNN을 통한 local feature 획득에 이점
- Transformer - Self Attention을 사용하여 global feature 획득에 이점
논문을 통하여 위의 두 이점이 TransUNet에서 어떻게 적용되었는지 살펴보고자 한다.
Method
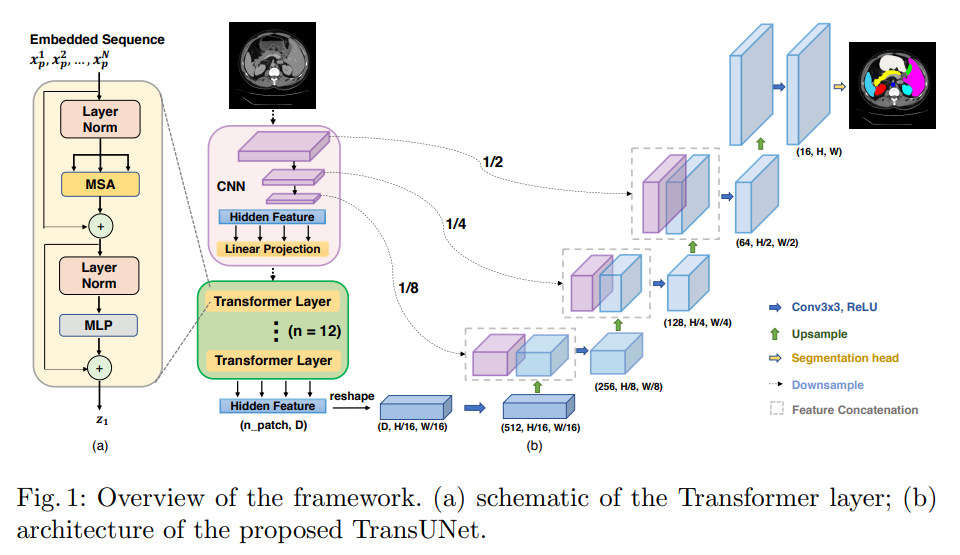
TransUNet의 architecture는 기본적으로 Unet의 구조를 따르며 아래와 같은 순서로 진행된다.
- CNN Block : Input data를 CNN을 통하여 local feature map을 생성
- Transformer Block : 생성된 feature map을 Transformer Encoder에 통과시켜 global feature를 생성
- Upsample Block: Upsample과정에서 skip connection을 통해 local & global feature를 반영
이제 모델의 간단한 흐름은 알았으니, 각 Block이 어떻게 진행되는지 자세히 살펴보기로 한다.
CNN Block
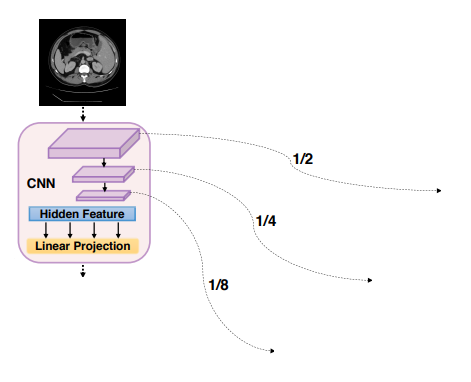
목적: CNN을 통하여 원래 크기의 1/2, 1/4, 1/8에 해당하는 local feature맵을 저장하는 것.
1. Input data를 받아 downsample을 진행한다.
- Input data : x ∈ R^(H*W*C) / H = H, W = W, C = input channel
2. downsample 단계마다 size는 1/2, channel은 2 배씩 영향을 받는다. (단, 첫 downsample에서만 output channel은 output channel을 받아서 사용한다.)
- 1 downsample : H = H , W = W, C = input channel -> H = 1/2 *H=, W = 1/2 *W, C = output channel
- 2 downsample : H = 1/2 *H=, W = 1/2 *W, C = output channel -> H = 1/4 *H=, W = 1/4 *W, C = 2*output channel
- 3 downsample : H = 1/4 *H=, W = 1/4 *W, C = 2*output channel -> H = 1/8 *H=, W = 1/8 *W, C = 4*output channel
- 4 downsample : H = 1/8 *H=, W = 1/8 *W, C = 4*output channel -> H = 1/16 *H=, W = 1/16 *W, C = 8*output channel
3. 각 downsample 단계마다 local feature map을 저장한다. (x1, x2, x3, x)
- xi : i번 downsample을 진행한 local feature map -> skip connection에 이용되는 데이터
- x: 4번 downsample을 진행한 local feature map으로, Transformer encoder에 이용
Transformer Block
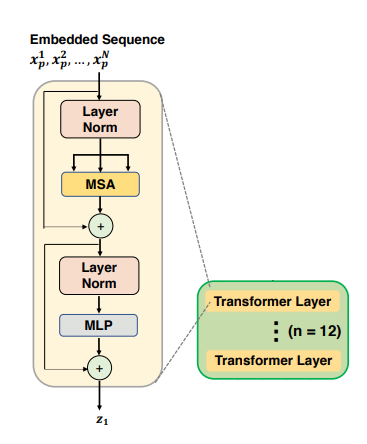
목적: CNN에서는 파악하기 힘든 global feature를 생성
1. 1/16크기의 x를 sequence of flattened 2D patches로 reshape
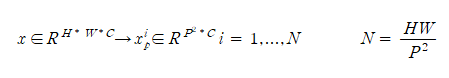
2. x_p에 위치정보를 추가하여 Z_0를 생성
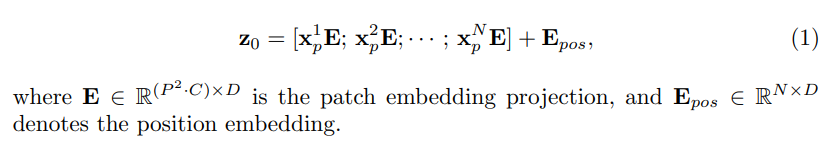
3. Z_0를 Transformer encoder에 통과시켜 Z_l를 생성
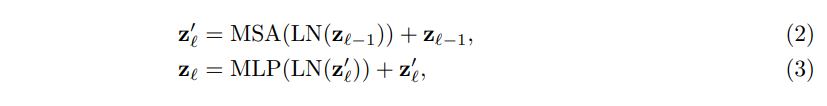
Upsample Block: Upsample
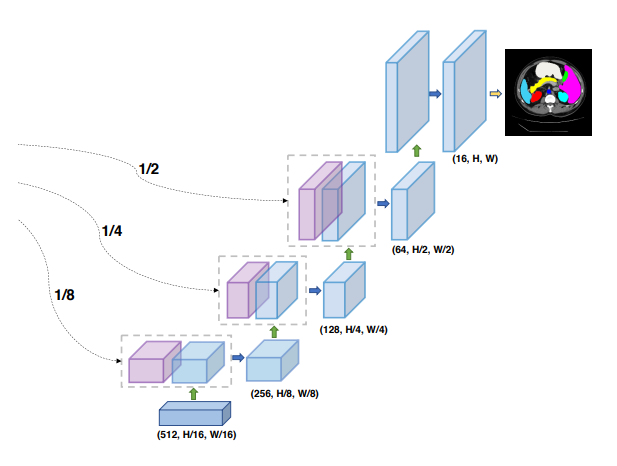
목적: global feature map과 local feature map을 원래의 H,W으로 회복.
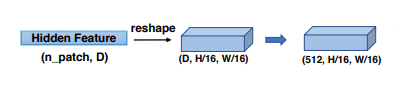
0. Transformer에서 나온 Z_l을 (D,H/16,W16)으로 reshape + D -> 512 channel로 convolution (downsample 부분)
1. x ∈ R^(512*1/16H*1/16W) 에 대하여 upsample을 진행
- 1 upsample : H = 1/16*H , 1/16*W = W, C = 512 -> H = 1/8 *H=, W = 1/8 *W, C = 128
- 2 upsample : H = 1/8*H , 1/8*W = W, C = 256 -> H = 1/4 *H=, W = 1/4 *W, C = 64
- 3 upsample : H = 1/4*H , 1/4*W = W, C = 128 -> H = 1/2 *H=, W = 1/2 *W, C = 32
- 4 upsample : H = 1/2*H , 1/2*W = W, C = 64 -> H = H=, W = W, C = 16
2. 1,2,3 upsample의 결과물을 x1,x2,x3와 channel에 대하여 concat을 진행 (skip connection)
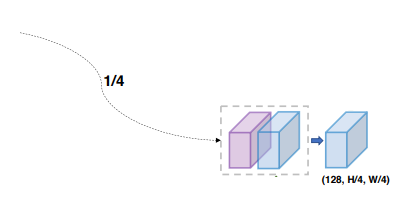
- upsample 1, x1 를 channel에 대하여 concat을 진행 -> H = 1/8*H , 1/8*W = W, C = 256
- upsample 2, x2 를 channel에 대하여 concat을 진행 -> H = 1/4*H , 1/4*W = W, C = 128
- upsample 3, x3 를 channel에 대하여 concat을 진행 -> H = 1/2*H , 1/2*W = W, C = 64
각 upsample 과정에서 channel에 대하여 합치기 때문에 upsample 이전 채널의 1/2이 되며, local feature map의 정보를 저장하게 된다.
3. 원하는 class의 segmentaion 된 image 획득
- upsample 4의 데이터를 이용하여 3*3 convolution을 진행
- (16,H,W)의 데이터를 원하는 class의 (class,H,W)의 데이터로 convolution을 진행
위와 같은 과정을 통하여, CNN을 통한 고해상도의 local feature map과 Transformer의 global feature map이 어떻게 segmentation에 적용되는지 알아보았다. 이제 TransUNet이 정말로 medical image segmentation에 효과적인지 저자의 비교실험결과를 통하여 살펴보고자 한다.
Experiments and Discussion
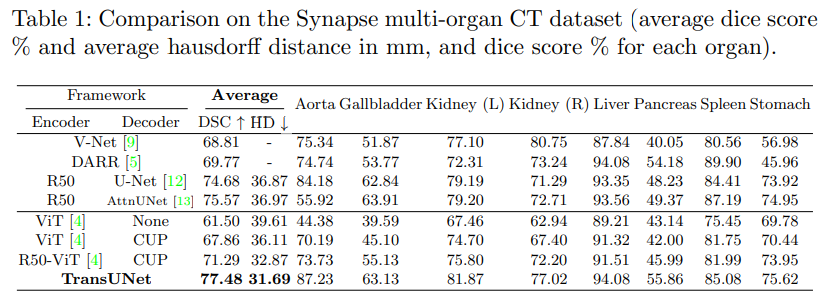
- 당시의 SOTA모델들과의 비교실험을 통해 TransUNet의 성능을 입증함.
- V-Net(3D segmentaion)과의 비교를 통해 3D보다도 좋은 성능을 보이는 것을 확인함.
- R20-ViT-CUP은 skip connection을 적용하지 않은 모델로 TransUNet과 DSC에서 6.19%의 성능 차이를 보임.
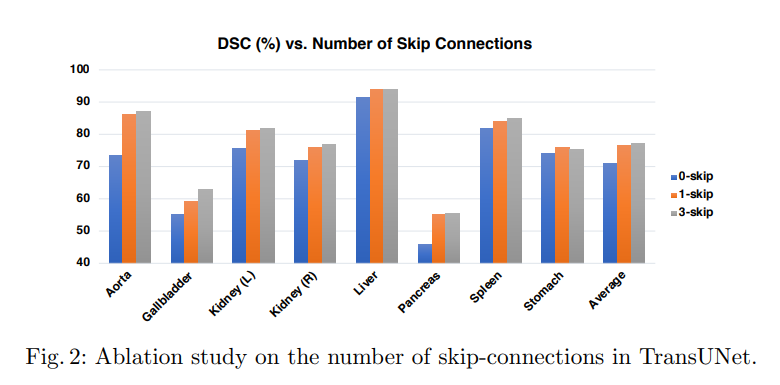
- skip connection의 중요도를 보여주는 지표로 0,1,3번의 skip에 대하여 3번 skip한 결과가 가장 우수하다고 보여짐.
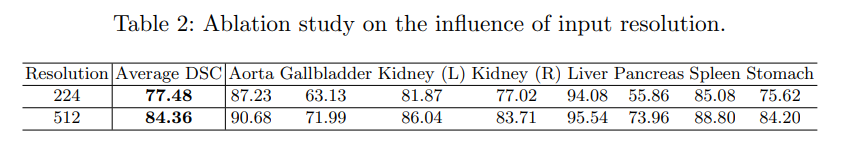
- input size에 대하여 224, 512를 비교한 지표로, size를 512로 하였을 때 224 대비 약 6%성능이 높은 것을 확인.
- 512의 경우 계산비용이 너무 늘어나 224에 비하여 비효율적임으로 저자는 224의 size를 채택
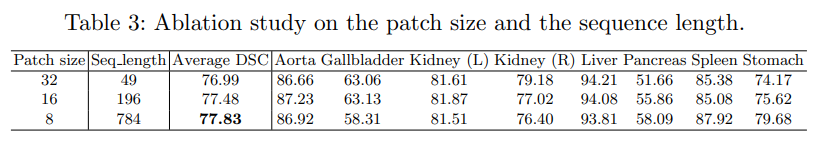
- patch size를 비교실험한 결과로 patch size가 작을수록 더 높은 성능을 보이는 것을 확인.
- 저자의 경우 16의 patch size를 채택
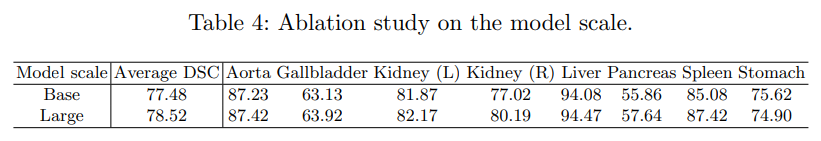

- Transformer encoder의 hyperparameters에 대하여 비교실험한 결과로 Large scale에서 더 좋은 성능을 보임.
- 모델의 계산비용의 문제로 저자는 Base scale을 채택함
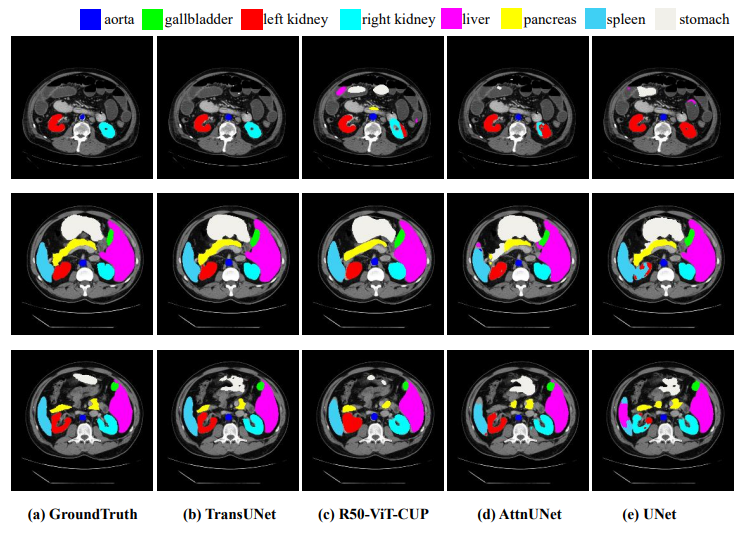
- visualization을 제시한 결과로, 각 model과 round-truth를 비교함